云舟生物科技(广州)股份有限公司品牌商
8 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0.5
- 1.5
- 0.5
- 3.5
公司新闻/正文
载体家小课堂 | 详解SARS-CoV-2的29种病毒蛋白
6220 人阅读发布时间:2021-07-05 16:33

详解SARS-CoV-2的29种病毒蛋白
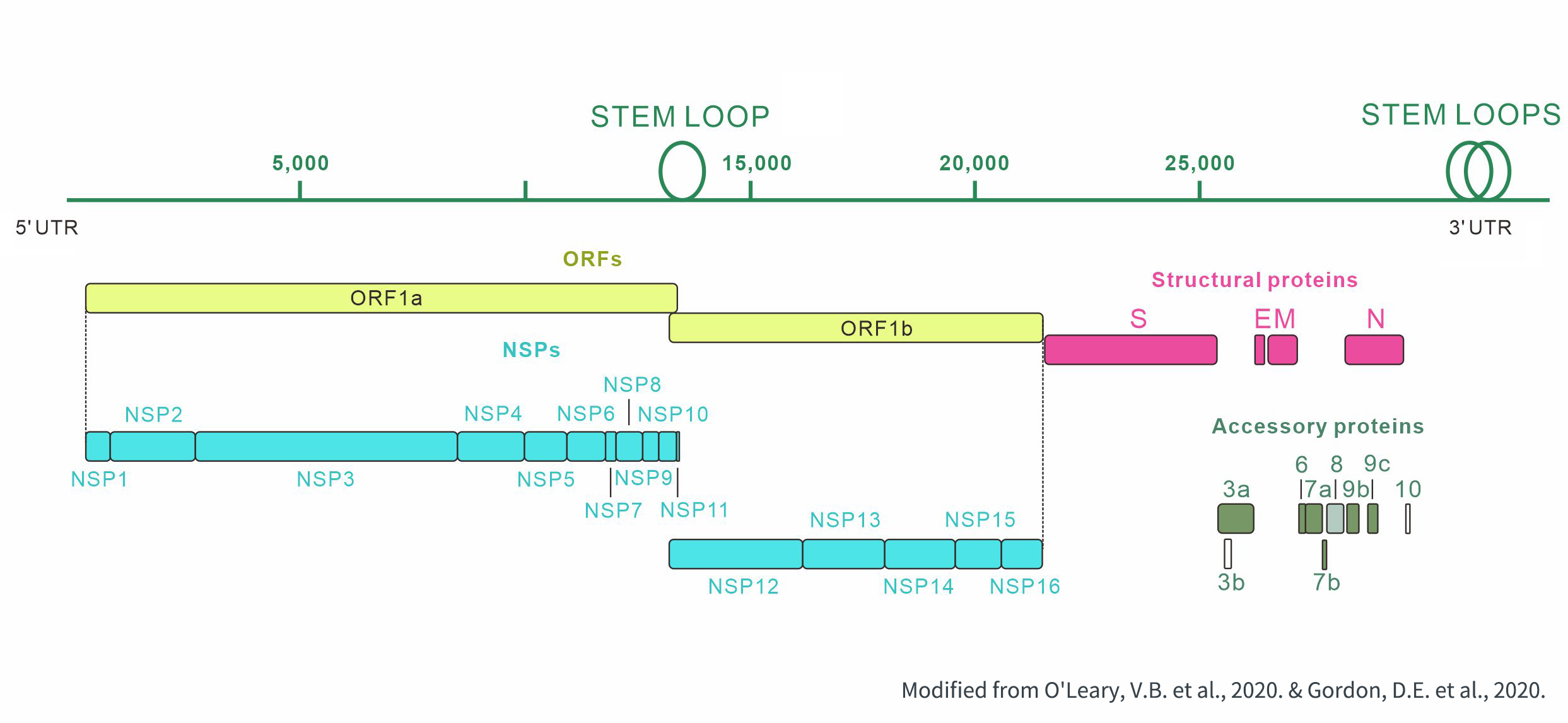
SARS-CoV-2基因组可翻译出的蛋白类型划分为3大类:非结构蛋白(Nonstructural protein)、结构蛋白(Structural protein)和辅助蛋白(Accessory protein)。其中非结构蛋白的ORF序列位于基因组上游,结构蛋白和辅助蛋白的ORF位于基因组下游。成熟的SARS-CoV-2主要由结构蛋白和病毒RNA基因组组成,非结构蛋白和辅助蛋白在病毒进入细胞后通过病毒RNA基因组的翻译产生。
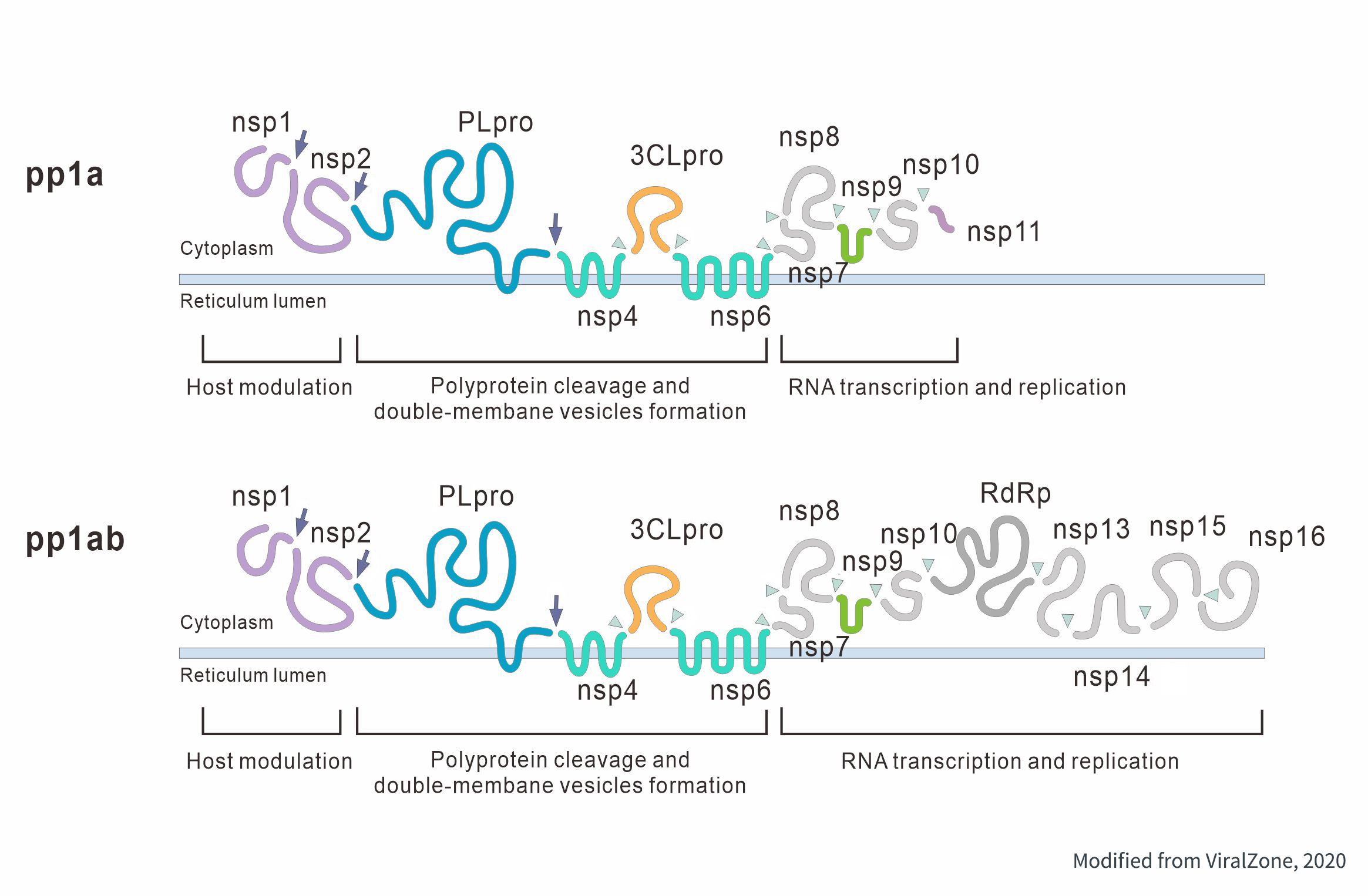
SARS-CoV-2的多聚蛋白结构示意图
(蓝色箭头:PLpro切割位点;灰色箭头:3CLpro切割位点)
非结构蛋白
由于非结构蛋白对应的RNA基因组上存在一个茎环结构(Stem loop),核糖体在翻译时通过发生阅读框滑动(Frameshift)形成两个开放阅读框:ORF1a和ORF1b。对应的翻译产物为两种多聚蛋白(Polyprotein)形式的多肽:pp1a和pp1ab。多聚蛋白自身没有功能,然而NSP3和NSP5可以从多聚蛋白上自水解并脱离,进一步将pp1a和pp1ab切割为成熟的非结构蛋白。以下为这些非结构蛋白的功能注释[2]:
NSP1
NSP1可以阻碍核糖体结合宿主mRNA,关闭宿主自身蛋白的合成路径。
NSP2
可直接结合宿主的抗增殖蛋白1和(Prohibitin 1,PHB1)和抗增殖蛋白2(Prohibitin 2,PHB2)。研究推测NSP2通过劫持PHB1和PHB2抑制细胞的自噬防御机制并破坏线粒体功能以有帮助病毒复制[3]。
NSP3
病毒的复制转录复合物中分子量最大的蛋白。该蛋白具有多个保守结构,其中木瓜样蛋白酶(PLpro)结构域帮助NSP3从多聚蛋白上水解释放,并对多聚蛋白N端造成切割,产生独立有功能的NSP1和NSP2。此外,NSP3还充当骨架蛋白的角色,与NSP4、NSP6相互作用,促进具有双层膜囊泡(Double-membrane vesicle, DMV)结构的病毒复制细胞器(Viral replication organelle)形成[4]。
NSP4、NSP6
跨膜蛋白,利用内质网的质膜介导DMV生成。
NSP5
3CL蛋白酶(3CLpro),也称为主蛋白酶。3CLpro通过自水解将自身从多聚蛋白释放,并对多聚蛋白上的NSP4到NSP16对应区域进行切割使其各自形成功能性蛋白。用于治疗HIV感染的蛋白酶靶向药物Lopinavir和Ritonavir,已被用于研究是否可同样有效作用于SARS-CoV-2的蛋白酶[5]。
NSP7、NSP8
NSP7与NSP8组成十六聚体复合物,充当NSP12复制病毒RNA时的引物酶。
NSP9
二聚体形式的单链RNA结合蛋白,稳定病毒RNA。
NSP10
激活NSP16的2’-O-甲基转移酶活性。NSP10同时也是稳定NSP14核酸外切酶结构域的辅因子。
NSP11
功能未知
NSP12
RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase, RdRp),拥有NSP7和NSP8两个辅因子,复制病毒RNA。Remdesivir即是以病毒RdRp为靶标而设计的COVID-19治疗药物[6]。
NSP13
RNA解旋酶,NSP12复制病毒RNA时解开双链RNA所需。
NSP14
一种双功能的酶,具有3’-5‘核酸外切酶结构域(ExoN),增强病毒复制RNA时的校对能力。NSP14也具有N7-甲基转移酶活性,修饰病毒RNA的5’鸟苷酸帽子,避免病毒RNA被细胞识别为外源RNA而被降解。
NSP15
RNA核酸内切酶,可以降解病毒的RNA碎片,减少其诱导宿主细胞启动抗病毒防卫机制的可能。
NSP16
一种2’-O-甲基转移酶,对病毒RNA的5’帽子进行甲基化修饰,避免病毒RNA被细胞识别为外源RNA而被降解。
结构蛋白
SARS-CoV-2是利用宿主的磷脂分子组装的包膜病毒,此外拥有四种结构蛋白:S、E、M、N。冠状病毒表面的花冠外形来源于其包膜上S蛋白刺突结构。以下为这些结构蛋白的功能注释[2]:
Spike (S)
SARS-CoV-2的表面刺突蛋白,识别宿主细胞表面ACE受体,介导病毒包膜与细胞膜融合,致使病毒RNA基因组进入细胞内。S蛋白突变与SARS-CoV-2变异株的感染能力增强密切相关,因此也是大量中和抗体的研究靶点。
Envelope (E)
E蛋白是病毒包膜上的序列高度保守的跨膜蛋白。研究认为E蛋白是一种病毒孔蛋白(Virion),可以扰乱细胞的离子浓度稳态,激活NLRP3炎性小体的生成[7]。
Membrane (M)
与病毒颗粒的组装有关。M蛋白与N蛋白的结合使得病毒RNP保持结构稳定[8]。
Nucleocapsid (N)
病毒翻译产生的数量最多的蛋白类型,序列在冠状病毒中高度保守。N蛋白结合病毒RNA,组装病毒时负责将线性的病毒基因组RNA包裹成致密的螺旋形式的核糖核蛋白(Ribonucleoprotein,RNP)复合体。
辅助蛋白
除了编码非结构蛋白和结构蛋白外,SARS-CoV-2基因组还编码9种辅助蛋白。研究表明辅助蛋白基因对冠状病毒复制是非必须的,但是其中一些辅助蛋白可以帮助病毒对抗宿主的病毒防御机制。以下为这些辅助蛋白的相关注释[2]:
ORF3a
作为离子通道的病毒孔蛋白,扰乱细胞的钙离子浓度稳态,过度激活NLRP3炎性小体的生成,最终引发细胞因子风暴[9]。
ORF3b
研究表明ORF3b抑制干扰素IFN-I的生成[10]。
ORF6
阻断干扰素信号通路中的STAT1转录因子进入细胞核,从而阻碍参与固有免疫应答相关的蛋白表达。
ORF7a
ORF7a劫持细胞膜上的骨髓基质抗原2(Bone Marrow Stromal Antigen 2,BST-2),减弱其正常功能。BST-2又称为Tetherin蛋白,可以物理性地将正在出芽的包膜病毒拴在细胞膜上,减少感染细胞中的病毒颗粒释放[11]。
ORF7b
分布于高尔基体上的III型跨膜蛋白,具体功能未知。
ORF8
SARS-CoV-2的ORF8不具备任何特定的结构域或者模 体,功能有待研究。
ORF9b、ORF9c
该蛋白的基因序列重叠于核衣壳蛋白N的基因序列,蛋白功能待进一步研究。
ORF10
研究表明该蛋白对SARS-CoV2-2的复制是非必要的,具体功能有待研究[12]。
重组病毒蛋白的人工表达
载体家拥有全面的重组蛋白表达与纯化技术平台,包括大肠杆菌表达系统、酵母表达系统、哺乳动物细胞表达系统和杆状病毒-昆虫细胞表达系统,可满足多种类型的病毒蛋白研究需求。
使用大肠杆菌表达系统
大肠杆菌E. coli仍然是当前最广泛使用的重组蛋白表达系统。该系统的表达载体通常采用T7噬菌体启动子,在含有T7 RNA聚合酶的大肠杆菌中驱动基因高效表达。在T7启动子下游引入乳糖操纵子增加表达调控功能,在添加IPTG后才会诱导重组蛋白的产生。由于大肠杆菌培养成本低、易放大、周期短,目的基因表达水平高,特别是对于小分子量的病毒蛋白,其表达产物经过纯化结晶后常用于蛋白质三维结构研究。
使用哺乳动物细胞表达系统
哺乳动物细胞如HEK293,作为一种真核蛋白表达系统,使用克隆有目的基因的基因表达质粒载体转染细胞即可实现重组蛋白表达。利用哺乳动物细胞表达重组病毒蛋白最大的优势在于该系统可以尽可能模拟病毒蛋白在被感染细胞中的实际分子生物学行为。哺乳动物细胞提供完整的翻译后修饰环境与蛋白转运链,有助于重组蛋白的正确折叠与正常生物学功能行使。哺乳动物细胞表达系统是当今研究病毒蛋白功能的常用工具,特备是在病毒-宿主蛋白互作、蛋白质组学分析与药物/抗体靶点研究方面有着重要应用。
使用杆状病毒-昆虫细胞表达系统
杆状病毒-昆虫细胞表达系统同为真核蛋白表达系统,对比哺乳动物细胞更适合需要大量表达的应用。该系统的重组蛋白经过细胞分泌,可以在培养基中获得非常高密度的重组蛋白,有利于分离纯化。得益于昆虫细胞可以正常地为重组蛋白进行磷酸化修饰,杆状病毒-昆虫细胞表达系统常用于表达激酶。该系统的另一个优势是无需使用动物源试剂,在避免BSE方面安全性高,因此被广泛用于临床研究,比如大量表达用于抗体研究的冠状病毒刺突蛋白以及抗体本身的生产。
参考文献
[1] Robson F, Khan KS, Le TK, Paris C, Demirbag S, Barfuss P, Rocchi P, Ng WL. Coronavirus RNA Proofreading: Molecular Basis and Therapeutic Targeting. Mol Cell. 2020 Sep 3;79(5):710-727.
[2] O'Leary VB, Dolly OJ, Höschl C, Černa M, Ovsepian SV. Unpacking Pandora From Its Box: Deciphering the Molecular Basis of the SARS-CoV-2 Coronavirus. Int J Mol Sci. 2020 Dec 31;22(1):386.
[3] Garvin MR, T Prates E, Pavicic M, Jones P, Amos BK, Geiger A, Shah MB, Streich J, Felipe Machado Gazolla JG, Kainer D, Cliff A, Romero J, Keith N, Brown JB, Jacobson D. Potentially adaptive SARS-CoV-2 mutations disCoVered with novel spatiotemporal and explainable AI models. Genome Biol. 2020 Dec 23;21(1):304.
[4] Zhang J, Lan Y, Sanyal S. Membrane heist: Coronavirus host membrane remodeling during replication. Biochimie. 2020;179:229-236.
[5] Meini S, Pagotto A, Longo B, Vendramin I, Pecori D, Tascini C. Role of Lopinavir/Ritonavir in the Treatment of CoVid-19: A Review of Current Evidence, Guideline Recommendations, and Perspectives. J Clin Med. 2020;9(7):2050.
[6] Frediansyah A, Nainu F, Dhama K, Mudatsir M, Harapan H. Remdesivir and its antiviral activity against COVID-19: A systematic review. Clin Epidemiol Glob Health. 2021;9:123-127.
[7] Singh Tomar PP, Arkin IT. SARS-CoV-2 E protein is a potential ion channel that can be inhibited by Gliclazide and Memantine. Biochem Biophys Res Commun. 2020;530(1):10-14.
[8] Schoeman D, Fielding BC. Coronavirus envelope protein: current knowledge. Virol J. 2019 May 27;16(1):69.
[9] Freeman TL, Swartz TH. Targeting the NLRP3 Inflammasome in Severe COVID-19. Front Immunol. 2020;11:1518.
[10] Yoriyuki Konno, Izumi Kimura, Keiya Uriu, Masaya Fukushi, Takashi Irie, Yoshio Koyanagi, Daniel Sauter, Robert J. Gifford, So Nakagawa, Kei Sato, SARS-CoV-2 ORF3b Is a Potent Interferon Antagonist Whose Activity Is Increased by a Naturally Occurring Elongation Variant,Cell Reports, Volume 32, Issue 12, 2020, 108185.
[11] Taylor JK, Coleman CM, Postel S, et al. Severe Acute Respiratory Syndrome Coronavirus ORF7a Inhibits Bone Marrow Stromal Antigen 2 Virion Tethering through a Novel Mechanism of Glycosylation Interference. J Virol. 2015;89(23):11820-11833.
[12] Pancer K, Milewska A, Owczarek K, Dabrowska A, Kowalski M, Łabaj PP, Branicki W, Sanak M, Pyrc K. The SARS-CoV-2 ORF10 is not essential in vitro or in vivo in humans. PLoS Pathog. 2020 Dec 10;16(12):e1008959.
[13] Gordon DE, Jang GM, Bouhaddou M, et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature. 2020;583(7816):459-468.
[14] https://viralzone.expasy.org/9076
云舟生物科技(广州)有限公司
020-28069042
service@vectorbuilder.cn